Tutorial analisi spettri e correlazioni¶
Introduzione¶
Questo quaderno si inserisce nel materiale didattico che verrà fornito agli studenti del secondo modulo del corso di laboratorio di fisica 3A (per gli amici Labo 4).
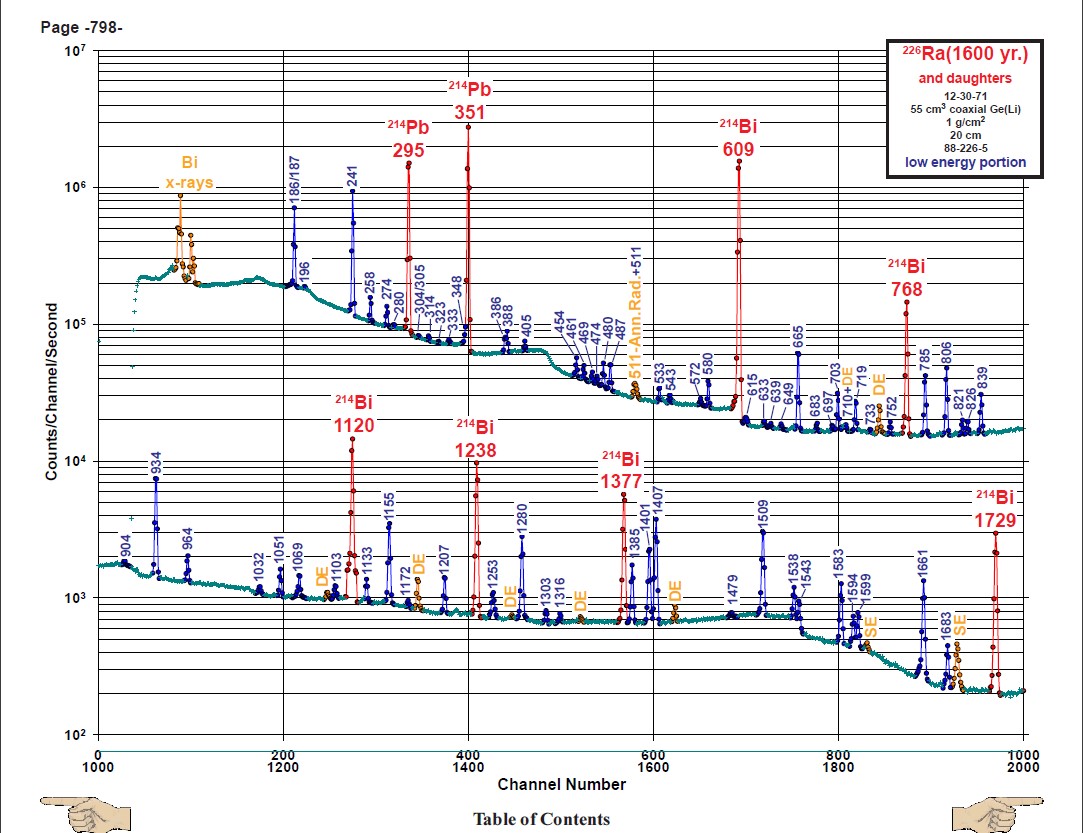
Abbiamo deciso di iniziare analizzando lo spettro del $^{226}$Ra, che è uno degli esempi più delicati e completi con cui avrete a che fare.
Nel caso durante il corso nascessero esigenze particolari riguardo a qualche dubbio nell'effettuare una certa procedura, oppure vi trovaste a dover lavorare con dati in formati strani, esattamente come durante lo scorso semestre, potete contattarmi e d'accordo con gli altri tutor e con i docenti, vedrò di venirvi incontro nel miglior modo possibile. La mia mail è scarsi@studenti.uninsubria.it
Scopo¶
In questo quaderno verranno presentati alcuni spunti necessari per poter costruire istogrammi relativi a spettri, fittarne i picchi e costruire istogrammi bidimensionali per studiare le correlazioni.
Il dataset utilizzato uno di quelli relativi all'analisi della spettroscopia gamma, ma le medesime tecniche si applicano ad un generico contesto in cui si hanno dei rivelatori da cui, sotto certe condizioni di trigger, viene estratto il massimo della waveform e si deve valutare la distribuzione dei massimi (come per esempio nel contesto della spettroscopia alpha).
Strumenti¶
L'analisi viene effettuata utilizzando Python 3 ed in particolare moduli numpy, matplotlib e scipy per la gestione e la visualizzazione di dati numerici.
Setup sperimentale e contesto¶
Il setup sperimentale è costituito da due tubi fotomoltiplicatori (PMT) connessi ad una catena elettronica di amplificazione e shaping. Ogni qualvolta almeno uno dei due PMT è sopra una soglia prestabilita, vengono acquisiti i massimi delle forme d'onda di ciascun canale.

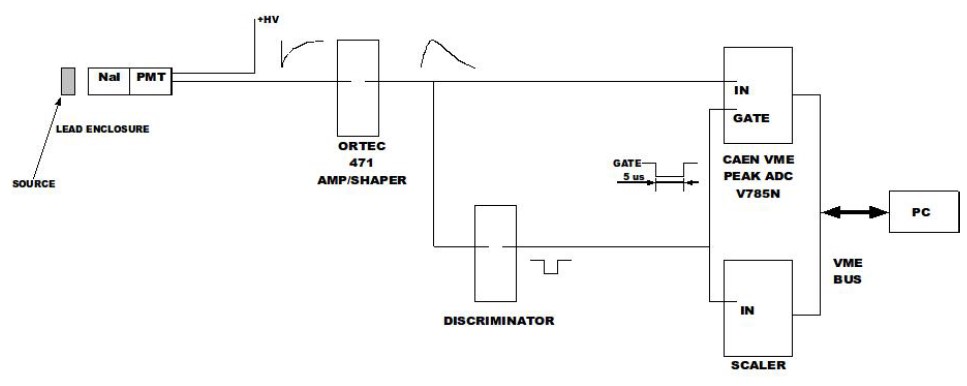
Potrebbe essere utile avere qualche informazione sul discriminatore...

Formato dei dati¶
I dati sono dei files di testo (in formato ASCII): ogni riga corrisponde ad un evento (ovvero ogni volta che almeno uno dei due PMT ha avuto valore sopra soglia), mentre ogni colonna contiene una certa quantità. Aprendo un file, esso ha la forma
...
001480064955 591 87 66 106
001480064955 130 6 4095 69
001480064955 235 92 66 106
001480064955 304 90 66 106
001480064955 858 87 66 107
001480064955 257 90 66 106
001480064955 249 84 66 106
001480064955 3717 85 68 106
001480064955 2498 75 69 107
...dove la prima colonna rappresenta il tempo unix) (ovvero il numero di secondi trascorsi dalla mezzanotte del 1° Gennaio 1970) mentre le successive quattro colonne rappresentano il massimo del segnale per ciascun canale. I due PMT sono connessi ai primi due canali. Poiché l'analisi è perfettamente equivalente, noi ci soffermeremo solamente sul primo canale.
Analisi dei dati¶
Iniziamo caricando (import) i moduli che verranno utulizzati durante l'analisi e definendo un alias per comodità nel richiamarli.