Tutorial analisi spettri e correlazioni¶
Introduzione¶
Questo quaderno si inserisce nel materiale didattico che verrà fornito agli studenti del secondo modulo del corso di laboratorio di fisica 3A (per gli amici Labo 4).
Ho scelto di ripercorrere l'analisi di un esperimento obbligatorio lo scorso anno ma facoltativo quest'anno poiché al suo interno contiene tutte le tecniche da utilizzare nelle altre attività ma al tempo stesso non fornisce un template già pronto da utilizzare come una black box per analizzare in automatico i dati raccolti.
Nel caso durante il corso nascessero esigenze particolari riguardo a qualche dubbio nell'effettuare una certa procedura, oppure vi trovaste a dover lavorare con dati in formati strani, esattamente come durante lo scorso semestre, potete contattarmi e d'accordo con gli altri tutor e con i docenti, vedrò di venirvi incontro nel miglior modo possibile. La mia mail è scarsi@studenti.uninsubria.it
Scopo¶
In questo quaderno verranno presentati alcuni spunti necessari per poter costruire istogrammi relativi a spettri, fittarne i picchi e costruire istogrammi bidimensionali per studiare le correlazioni.
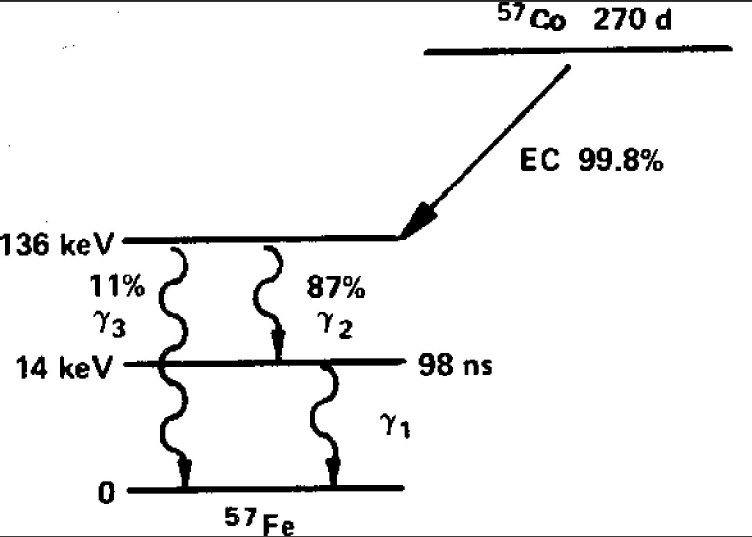
Il dataset utilizzato è quello relativo all'analisi della lifetime del $^{57}$Co (che quest'anno risulta essere una attività a scelta), ma le medesime tecniche si applicano ad un generico contesto in cui si hanno dei rivelatori da cui, sotto certe condizioni di trigger, viene estratto il massimo della waveform e si deve valutare la distribuzione dei massimi (come per esempio nel contesto della spettroscopia gamma, di cui vedremo un assaggio nell'esercitazione).
Strumenti¶
L'analisi viene effettuata utilizzando Python 3 ed in particolare moduli numpy, matplotlib e scipy per la gestione e la visualizzazione di dati numerici.
Setup sperimentale e contesto¶
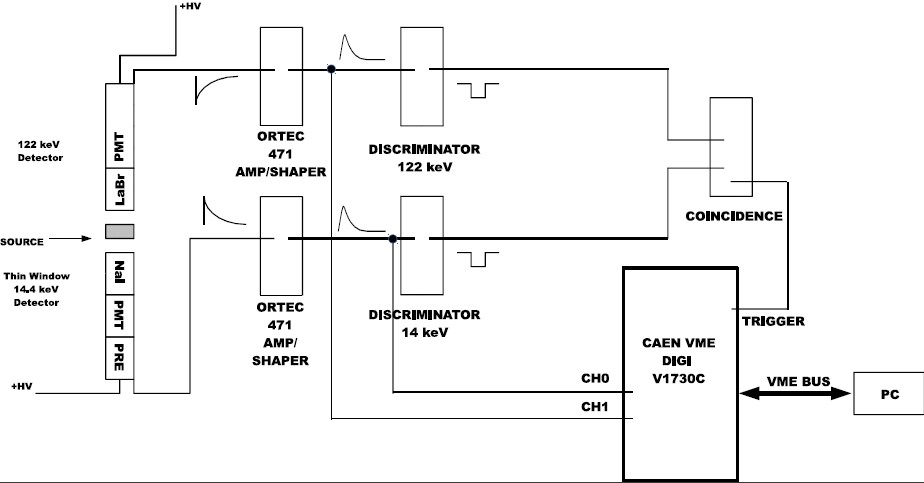
Il setup sperimentale è costituito da due tubi fotomoltiplicatori (PMT) connessi ad una catena elettronica di amplificazione e shaping. Ogni qualvolta almeno uno dei due PMT è sopra una soglia prestabilita, vengono acquisiti i massimi delle forme d'onda di ciascun canale.
Lo stato eccitato del $^{57}$Fe può decadere o emettendo un fotone da 136 keV (parte sx dello schema) che può venir rivelato da un PMT oppure emettendo in successione due fotoni da 122 keV e 14keV, che possono venir rivelati ciascuno da uno dei due PMT.
Formato dei dati¶
I dati sono dei files di testo (in formato ASCII): ogni riga corrisponde ad un evento (ovvero ogni volta che almeno uno dei due PMT ha avuto valore sopra soglia), mentre ogni colonna contiene una certa quantità. Aprendo un file, esso ha la forma
...
00000022 1556869569 00117 00479 02536 00924
00000023 1556869569 09368 00732 03898 00972
00000024 1556869569 00008 00054 11311 01006
00000025 1556869569 00002 00051 10155 01002
00000026 1556869569 00178 00847 15232 00840
00000027 1556869569 00010 00083 15228 00914
00000028 1556869569 00000 00000 00003 00052
00000029 1556869569 00029 00773 15227 00876
00000030 1556869569 00763 00208 00002 00053
00000031 1556869569 00872 00208 00002 00057
00000032 1556869569 00081 00851 08307 01006
...dove la prima colonna rappresenta il numero dell'evento, la seconda colonna il tempo unix) (ovvero il numero di secondi trascorsi dalla mezzanotte del 1° Gennaio 1970) mentre le successive colonne rappresentano il massimo del segnale e il relativo tempo rispetto al trigger per ciascuno dei due PMT.
Analisi dei dati¶
Iniziamo caricando (import) i moduli che verranno utulizzati durante l'analisi e definendo un alias per comodità nel richiamarli.